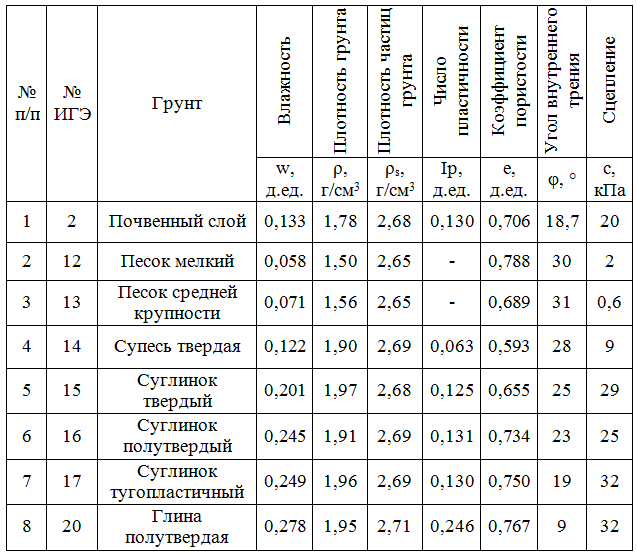
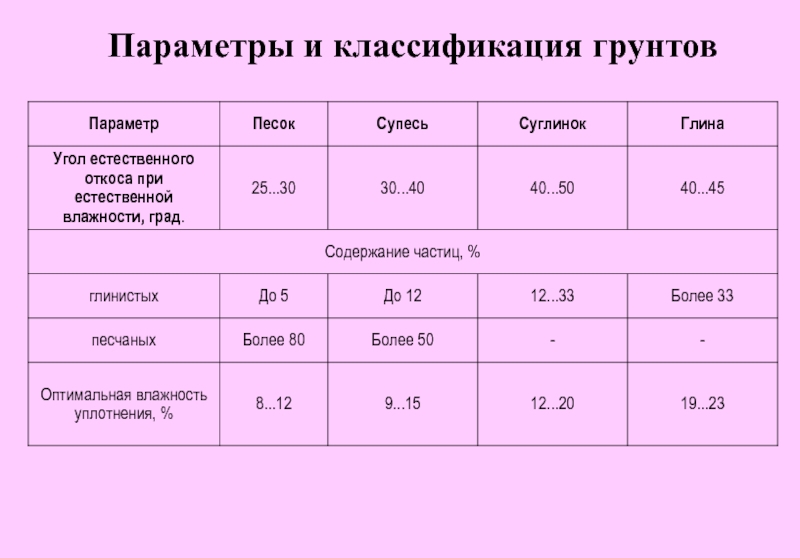
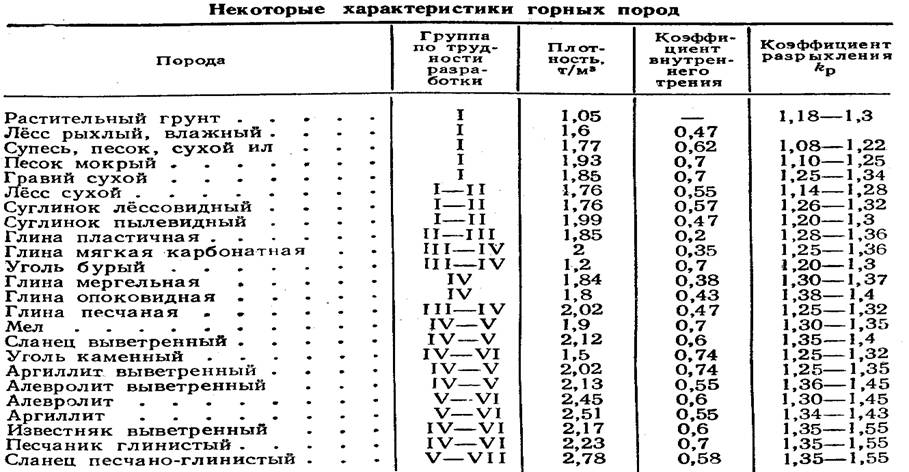
Полезная информация
Полезная информация- ГЛАВНАЯ
- БУРОВОЙ ИНСТРУМЕНТ
- Буровой инструмент
- Буры
- Буры лопастные
- Буры конусные
- Буры ковшевые
- Буры колонковые
- Буры траверсные
- Буры перовые
- Буры шнековые
- Короткошнековые на плоских резцах
- Короткошнековые на круглых резцах
- Долота
- Шарошечные
- Режущее оснащение
- Резцы плоские
- Резцы круглые
- Переходники
- Шарнирные
- Не шарнирные
- Шнеки
- Шнеки с частичной навивкой
- Шнеки непрерывные
- Шнеки телескопические
- Шнековые секции
- Шнеки короткие
- Забурники
- Забурники перовые
- Забурники плоские
- Карманы
- Карманы для плоских резцов
- Карманы для круглых резцов (цилиндрические)
- Карманы для круглых резцов (консольные)
- НАВЕСНОЕ ОБОРУДОВАНИЕ
- Гидровращатели (гидробуры)
- Малые приводы
- Средние приводы
- Крупные приводы
- Гидровращатели (гидробуры)
- ГАЛЕРЕЯ
- Адаптеры, Переходники
- Буры, Долота
- Вспомогательный инструмент
- Запчасти, Комплектующие
- Навесное оборудование
- Производство
- Резцы, Забурники, Карманы
- Сваи
- Склад
- Шнеки, Удлинители, Штанги
- ЗАПЧАСТИ И КОМПЛЕКТУЮЩИЕ
- РВД
- Металлоконструкции
- Моторы
- Насосы
- Гидроцилиндры
- ИНФОРМАЦИЯ
- Формулы заказов бурового инструмента
- Сертификаты
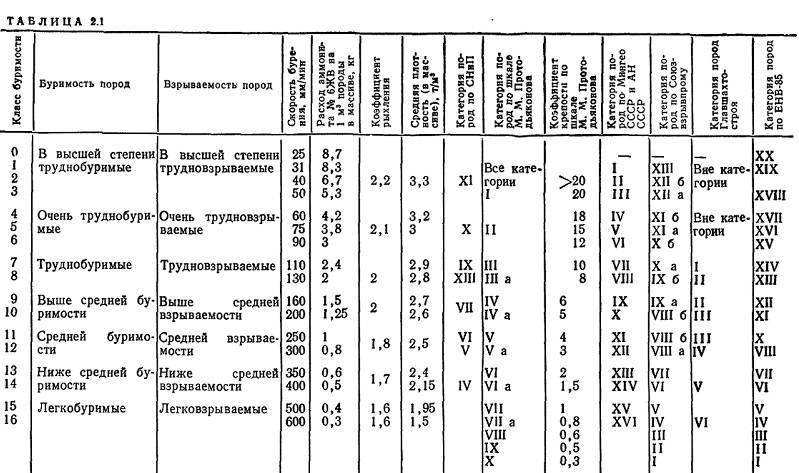
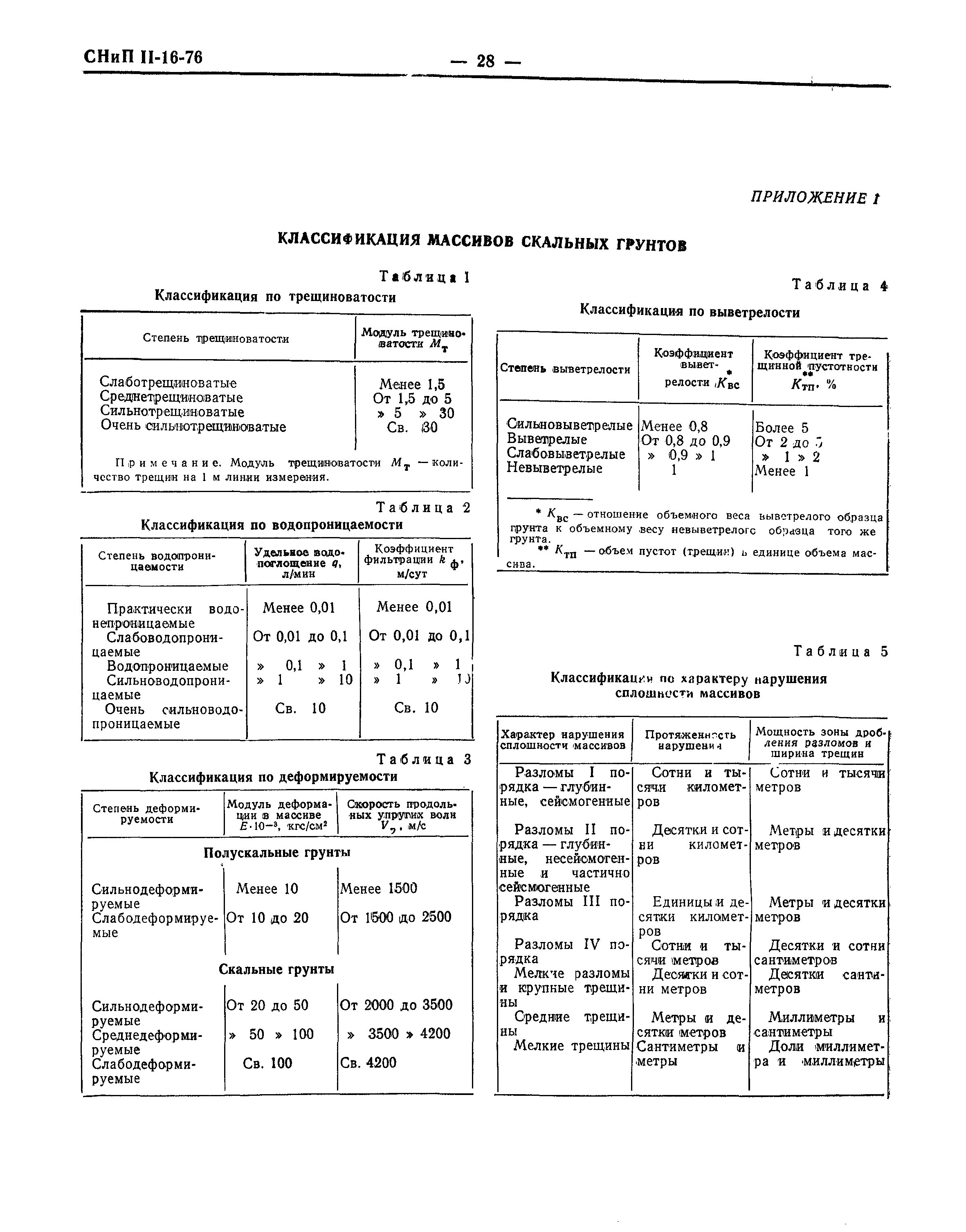
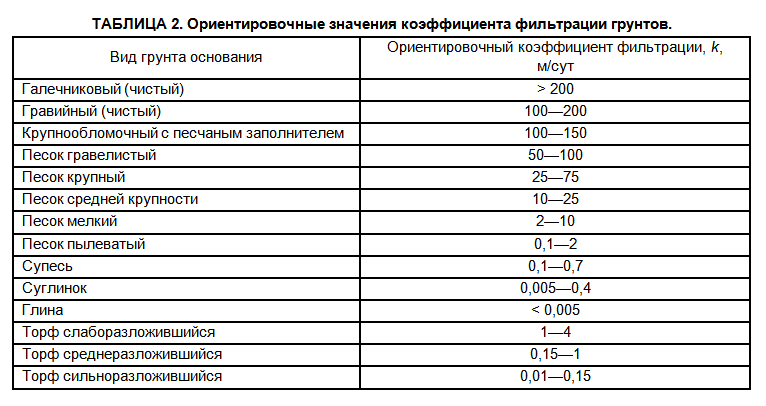
- Категория грунтов и пород буримости
- ГЕОГРАФИЯ ПОСТАВОК
- НОВОСТИ
Ваше ФИО*
Название компании*
ИНН*
Город*
Номер телефона*
Email*
Сообщение*
Файл
Классификация грунтов по льдистости и просадочности в I дорожно-климатической зоне
Разновидность грунта по просадочности при оттаивании | Льдистость1 грунта вечномерзлой толщи | Суммарная влажность грунтов деятельного слоя | |||
пески мелкие | пески пылеватые, супеси легкие | Супеси | торф | ||
| Непросадочный | Без ледяных включений (0 – 0,01) | Менее 0,18 | Менее 0,2 | Менее 0,2 | — |
Слабопросадочный | Малольдистый (0,01 – 0,1) | От 0,18 до 0,25 | От 0,2 до 0,4 | От 0,2 до 0,4 | Менее 2 |
Просадочный | Льдистый (0,1 – 0,4) | Св. | Св. 0,4 до 1,1 | От 2 до 12 | |
Сильнопросадочный | Сильнольдистый (0,4 – 0,6) | — | — | Св. 1,1 | Св. 12 |
Чрезмерно просадочный | С крупными включениями подземного льда (0,6 – 1,0) | — | — | “ 1,1 | “ 12 |
1 Отношение объема прослоек льда к объему
мерзлого грунта (с учетом включений
частиц льда).
Таблица 11
Разновидность грунтов по степени увлажнения
Разновидность грунтов | Влажность |
Недоувлажненные Нормальной влажности Повышенной влажности Переувлажненные | Менее 0,9 wo От 0,9 wo до wadm “ wadm “ wmax Св. w |
Примечание. wmax – максимально возможная влажность грунта при коэффициенте уплотнения 0,9.
Таблица 12
Допустимая влажность грунтов при уплотнении
Грунты | Допустимая влажность wadm в долях от оптимальной при требуемом коэффициенте уплотнения | |||
Св. | 1,0 – 0,98 | 0,95 | 0,90 | |
Пески пылеватые; супеси легкие крупные | 1,30 | 1,35 | 1,60 | 1,60 |
Супеси легкие и пылеватые | 1,20 | 1,25 | 1,35 | 1,60 |
Продолжение табл. 12
Грунты | Допустимая влажность wadm в долях от оптимальной при требуемом коэффициенте уплотнения | |||
Св. | 1,0 – 0,98 | 0,95 | 0,90 | |
Супеси тяжелые пылеватые; суглинки легкие и легкие пылеватые | 1,10 | 1,15 | 1,30 | 1,50 |
Суглинки тяжелые и тяжелые пылеватые, глины | 1,0 | 1,05 | 1,20 | 1,30 |
Продолжение табл. 12
Примечания:
1. При возведения насыпей из непылеватых
песков в летних условиях допустимая
влажность не ограничивается.
2. Настоящее ограничения не распространяются на насыпи, возводимые гидронамывом.
3. При возведении насыпи в зимних условиях влажность не должна, как правило, быть более 1,3 wo при песчаных и непылеватых супесчаных, 1,2 wo – при супесчаных пылеватых и суглинках легких и 1,1 wo – для других связных грунтов.
4. Величина допустимой влажности грунта может уточнятся с учетом технологических возможностей имеющихся в наличии конкретных уплотняющих средств в соответствии с нормами СНиП 3.06.03 – 85.
Таблица 13
Идентификация SNP и разработка маркерного анализа для высокопроизводительной селекции на устойчивость к цистообразующим нематодам сои
. 2015 18 апреля; 16 (1): 314. doi: 10.1186/s12864-015-1531-3.Цзы Ши 1 , Шиминг Лю 2 , Джеймс Ноэ 3 , Пракаш Арелли 4 , Халид Мексем 5 , Цзэнглу Ли 6
Принадлежности
- 1 Центр прикладных генетических технологий и отд.
 растениеводства и почвоведения, Университет Джорджии, Афины, Джорджия, 30602, США. [email protected].
растениеводства и почвоведения, Университет Джорджии, Афины, Джорджия, 30602, США. [email protected]. - 2 Департамент растений, почв и сельскохозяйственных систем, Университет Южного Иллинойса, Карбондейл, Иллинойс, 62901, США. [email protected].
- 3 Кафедра патологии растений, Университет Джорджии, Афины, Джорджия, 30602, США. [email protected].
- 4 USDA-ARS-SEA, Джексон, Теннесси, 38301, США. [email protected].
- 5 Департамент растений, почв и сельскохозяйственных систем, Университет Южного Иллинойса, Карбондейл, Иллинойс, 62901, США. [email protected].
- 6 Центр прикладных генетических технологий и отд.
 растениеводства и почвоведения, Университет Джорджии, Афины, Джорджия, 30602, США. [email protected].
растениеводства и почвоведения, Университет Джорджии, Афины, Джорджия, 30602, США. [email protected].
- PMID: 250
- PMCID: PMC4407462
- DOI: 10.1186/s12864-015-1531-3
Цзы Ши и др. Геномика BMC. .
Бесплатная статья ЧВК . 2015 18 апреля; 16 (1): 314. doi: 10.1186/s12864-015-1531-3.Авторы
Цзы Ши 1 , Шимин Лю 2 , Джеймс Ноэ 3 , Пракаш Арелли 4 , Халид Мексем 5 , Цзэнглу Ли 6
Принадлежности
- 1 Центр прикладных генетических технологий и отд.
 растениеводства и почвоведения, Университет Джорджии, Афины, Джорджия, 30602, США. [email protected].
растениеводства и почвоведения, Университет Джорджии, Афины, Джорджия, 30602, США. [email protected]. - 2 Департамент растений, почв и сельскохозяйственных систем, Университет Южного Иллинойса, Карбондейл, Иллинойс, 62901, США. [email protected].
- 3 Кафедра патологии растений, Университет Джорджии, Афины, Джорджия, 30602, США. [email protected].
- 4 USDA-ARS-SEA, Джексон, Теннесси, 38301, США. [email protected].
- 5 Кафедра растений, почв и сельскохозяйственных систем, Университет Южного Иллинойса, Карбондейл, Иллинойс, 62901, США. [email protected].
- 6 Центр прикладных генетических технологий и отд.
 растениеводства и почвоведения, Университет Джорджии, Афины, Джорджия, 30602, США. [email protected].
растениеводства и почвоведения, Университет Джорджии, Афины, Джорджия, 30602, США. [email protected].
- PMID: 250
- PMCID: PMC4407462
- DOI: 10.1186/s12864-015-1531-3
Абстрактный
Фон: Соевая цистообразующая нематода (SCN) является наиболее разрушительным с экономической точки зрения патогеном сои. Два локуса устойчивости, Rhg1 и Rhg4, в первую очередь способствуют устойчивости к расе 3 SCN у сои. Peking и PI 88788 являются двумя основными источниками резистентности к SCN, при этом Peking требуются аллели Rhg1 и Rhg4, а PI 88788 — только аллель Rhg1. Хотя маркеры простого повтора последовательности (SSR) были зарегистрированы для обоих локусов, они являются сцепленными маркерами и ограничены для применения в программах разведения из-за точности, производительности и стоимости методов обнаружения. Цели этого исследования заключались в разработке надежных анализов функциональных маркеров для высокопроизводительного отбора резистентности к SCN и дифференциации источников резистентности.
Хотя маркеры простого повтора последовательности (SSR) были зарегистрированы для обоих локусов, они являются сцепленными маркерами и ограничены для применения в программах разведения из-за точности, производительности и стоимости методов обнаружения. Цели этого исследования заключались в разработке надежных анализов функциональных маркеров для высокопроизводительного отбора резистентности к SCN и дифференциации источников резистентности.
Полученные результаты: Основываясь на последовательностях геномной ДНК 27 линий сои с известными фенотипами SCN, мы разработали анализ конкурентной аллель-специфической ПЦР (KASP) для двух однонуклеотидных полиморфизмов (SNP) из Glyma08g11490 для отбора аллеля устойчивости Rhg4. Более того, геномная ДНК Glyma18g02590 в локусе Rhg1 из 11 линий сои и кДНК Forrest, Essex, Williams 82 и PI 88788 были полностью секвенированы. Попарное выравнивание последовательностей выявило семь SNP/вставок/делеций (InDels), пять в 6-м экзоне и два в последнем экзоне. Используя те же 27 линий сои, мы идентифицировали один SNP, который можно использовать для выбора аллеля устойчивости Rhg1, и другой SNP, который можно использовать для дифференциации устойчивости к пекинскому и PI 88788 типам. Эти SNP-маркеры были подтверждены, и наблюдалась сильная корреляция между генотипами SNP и реакцией на SCN расы 3 с использованием панели из 153 линий сои, а также двухродительской популяции, рекомбинантных инбредных линий, полученных из F5 (RIL) из G00. -3213xLG04-6000.
Используя те же 27 линий сои, мы идентифицировали один SNP, который можно использовать для выбора аллеля устойчивости Rhg1, и другой SNP, который можно использовать для дифференциации устойчивости к пекинскому и PI 88788 типам. Эти SNP-маркеры были подтверждены, и наблюдалась сильная корреляция между генотипами SNP и реакцией на SCN расы 3 с использованием панели из 153 линий сои, а также двухродительской популяции, рекомбинантных инбредных линий, полученных из F5 (RIL) из G00. -3213xLG04-6000.
Выводы: Были идентифицированы три функциональных SNP-маркера (два для локуса Rhg1 и один для локуса Rhg4), которые могут предоставить информацию о генотипе для отбора устойчивости к SCN и дифференцировать Пекин от источника PI 88788 для большинства линий зародышевой плазмы. Были разработаны надежные анализы маркеров KASP SNP. В большинстве случаев использования одного или двух из этих маркеров достаточно для высокопроизводительной селекции с помощью маркеров растений, которые будут проявлять устойчивость к SCN.
Цифры
Рисунок 1
Схематический график показал положение…
Рисунок 1
На схематическом графике показано положение SNP/InDels в локусах Rhg1 и Rhg4 . А…
Рисунок 1 На схематическом графике показано положение SNP/InDels в локусах Rhg1 и Rhg4 . А . Два SNP идентифицированы в Glyma08g11490 в локусе Rhg4 . Б . 13 SNP/InDels, идентифицированных в трех генах в пределах блока размером 31,2 т.п.н. в локусе Rhg1 . Синие квадраты представляют экзоны, а черные линии представляют UTR и/или интроны. Координаты генома (база данных: Gmax_275_v2.0.softmasked) показаны под каждым SNP в скобках. Аллели SNP, выделенные черным, взяты из опубликованных последовательностей, а аллели SNP, выделенные красным, взяты из наших результатов секвенирования.
Рисунок 2
Позиции трех SNP в…
Рисунок 2
Положения трех SNP в Glyma18g02590 и Glyma08g11490 в локусах Rgh2 и Rhg4 ,…
фигура 2Положения трех SNP в Glyma18g02590 и Glyma08g11490 по локусам Rgh2 и Rhg4 соответственно. Номера GSM использовались для обозначения маркеров SNP. Синие квадраты представляют экзоны, а черные линии представляют интроны и UTR. Показаны координаты генома (база данных: Gmax_275_v2.0.softmasked) каждого SNP.
Рисунок 3
Анализ гаплотипов по Rhg1 и…
Рисунок 3
Анализ гаплотипов в локусах Rhg1 и Rhg4 с использованием данных чипа Soy50k SNP Infinium. …
…
в локусах Rhg1 и Rhg4 с использованием данных чипа Soy50k SNP Infinium. Были использованы двадцать семь интродуцентов растений сои и девять сортов: Вудрафф, Хартвиг, Боггс, Гордон, Брайан, G00-3880, Причард, Бедфорд и Карвер. А . Анализ гаплотипов в 9 лет0157 Rhg1 местонахождение. Аллели SNP в области Rhg1 на Chr18 сравнивали с пекинским и PI 88788 соответственно. На графике показана область размером 788 кбит/с (координата 1 266 210–2 054 952 на Chr18 на основе Gmax_275_v2.0.softmasked), состоящая из 82 SNP. Б . Анализ гаплотипов по локусу Rhg4 . Аллели SNP в области Rhg4 на Chr08 сравнивали с пекинским. Здесь показана область 1,5 Мбит/с (координата 6 903 359-8 399 514 на Chr08 на основе Gmax_275_v2.0. softmasked), состоящая из 82 SNP. Опорная линия показана темно-красным цветом. Красные квадраты представляют те же аллели, что и эталон, а зеленые квадраты представляют аллели, которые отличаются от эталона. Положение локуса и типы резистентности указаны.
Положение локуса и типы резистентности указаны.
Рисунок 4
Графики KASP трех SNP…
Рисунок 4
KASP-графики трех SNP для валидации и прецизионного генотипирования тестовой популяции. А…
Рисунок 4KASP-графики трех SNP для тестовой популяции для валидации и точного генотипирования. А . Графики KASP SNP для 153 линий сои, проведенных в 2011–2013 гг. Южными унифицированными тестами сои Министерства сельского хозяйства США с известными реакциями SCN расы 3. Б . Графики KASP SNP для RIL, производных от F 5 , из популяции, полученной из G00-3213 x LG04-6000.
Рисунок 5
Реакции расы3 на разные…
Рисунок 5
Реакции расы 3 на разные комбинации маркерных аллелей SNP для 150 F…
Рисунок 5 Реакции расы3 на различные комбинации аллелей маркера SNP для 150 F 5 семей G00-3213 x LG04-6000. Генотипы Rhg1 и Rhg4 представлены комбинациями аллелей маркеров SNP, GSM381, GSM383 и GSM191. Прямоугольники показывают медиану, от 25% до 75% данных, а усы простираются до минимума и максимума данных.
Генотипы Rhg1 и Rhg4 представлены комбинациями аллелей маркеров SNP, GSM381, GSM383 и GSM191. Прямоугольники показывают медиану, от 25% до 75% данных, а усы простираются до минимума и максимума данных.
См. это изображение и информацию об авторских правах в PMC
Похожие статьи
- Геномный филогенетический анализ и разработка маркеров для селекции на устойчивость к цистообразующим нематодам сои следующего поколения.
Кадам С., Выонг Т.Д., Цю Д., Мейнхардт К.Г., Сонг Л., Дешмукх Р., Патил Г., Ван Дж., Валлиодан Б., Скабу А.М., Шеннон Д.Г., Нгуен Х.Т. Кадам С. и др. Растениевод. 2016 Январь; 242: 342-350. doi: 10.1016/j.plantsci.2015.08.015. Epub 2015 28 августа. Растениевод. 2016. PMID: 26566850
- Полногеномный анализ ассоциации указывает на дополнительные основные области генома, придающие устойчивость к цистообразующим нематодам сои ( Heterodera glycines Ichinohe).

Tran DT, Steketee CJ, Boehm JD Jr, Noe J, Li Z. Тран Д.Т. и др. Фронт завод науч. 2019 10 апр; 10:401. doi: 10.3389/fpls.2019.00401. Электронная коллекция 2019. Фронт завод науч. 2019. PMID: 31031779Бесплатная статья ЧВК.
- Генетическое разнообразие локусов rhg1 и Rhg4 в дикой сое, устойчивой к соевой нематоде расы 3.
Юань С.П., Ван Ю.Дж., Чжао Х.К., Чжан Л., Ван Ю.М., Лю С.Д., Чжун С.Ф., Донг Ю.С. Юань С.П. и др. Жене Мол Рез. 2016 10 июня; 15 (2). doi: 10.4238/gmr.15027386. Жене Мол Рез. 2016. PMID: 27323148
- Достижения в селекции, генетике и геномике для устойчивости к трем видам нематод в сое.
Ким К.С., Выонг Т.Д., Цю Д., Роббинс Р.Т., Гровер Шеннон Дж., Ли З., Нгуен Х.Т. Ким К.
 С. и др.
Теория Appl Genet. 2016 декабрь; 129(12):2295-2311. doi: 10.1007/s00122-016-2816-x. Epub 2016 28 октября.
Теория Appl Genet. 2016.
PMID: 27796432
Обзор.
С. и др.
Теория Appl Genet. 2016 декабрь; 129(12):2295-2311. doi: 10.1007/s00122-016-2816-x. Epub 2016 28 октября.
Теория Appl Genet. 2016.
PMID: 27796432
Обзор. - Интеграция локусов устойчивости к синдрому внезапной смерти в геноме сои.
Chang HX, Roth MG, Wang D, Cianzio SR, Lightfoot DA, Hartman GL, Chilvers MI. Чанг Х.Х. и др. Теория Appl Genet. 2018 Апрель; 131 (4): 757-773. doi: 10.1007/s00122-018-3063-0. Epub 2018 12 февраля. Теория Appl Genet. 2018. PMID: 29435603 Обзор.
Посмотреть все похожие статьи
Цитируется
- Разработка и внедрение эффективного рабочего процесса генотипирования для ускорения улучшения качества кукурузы в развивающихся странах.
Офорнедо К., Менкир А.
 , Бабалола Д., Гедил М.
Оффорнедо К. и др.
Ворота открыты рез. 2022 3 августа; 6:3. doi: 10.12688/gatesopenres.13338.3. Электронная коллекция 2022.
Ворота открыты рез. 2022.
PMID: 37398911
Бесплатная статья ЧВК.
, Бабалола Д., Гедил М.
Оффорнедо К. и др.
Ворота открыты рез. 2022 3 августа; 6:3. doi: 10.12688/gatesopenres.13338.3. Электронная коллекция 2022.
Ворота открыты рез. 2022.
PMID: 37398911
Бесплатная статья ЧВК. - Локусы количественных признаков и геноспецифические маркеры, ассоциированные с устойчивостью к соевой нематоде HG типа 2.5.7.
Лу Х, Тораби С., де Лима Пассианотто А.Л., Велаки Т., Эскандари М. Лу X и др. Мол Порода. 2022 сен 29;42(10):62. doi: 10.1007/s11032-022-01330-8. Электронная коллекция 2022 окт. Мол Порода. 2022. PMID: 37313012
- Элитные варианты гермоплазмы для селекции сои.
Li D, Zhang Z, Gao X, Zhang H, Bai D, Wang Q, Zheng T, Li YH, Qiu LJ. Ли Д и др. Мол Порода. 2023 2 мая; 43(5):37.
 doi: 10.1007/s11032-023-01378-0. Электронная коллекция 2023 май.
Мол Порода. 2023.
PMID: 37312749
doi: 10.1007/s11032-023-01378-0. Электронная коллекция 2023 май.
Мол Порода. 2023.
PMID: 37312749 - Идентификация устойчивых источников из Glycine max против цистообразующих нематод сои.
Лиан Ю, Юань М, Вэй Х, Ли Дж, Дин Б, Ван Дж, Лу В, Кох Г. Лиан Ю и др. Фронт завод науч. 2023 7 марта; 14:1143676. дои: 10.3389/fpls.2023.1143676. Электронная коллекция 2023. Фронт завод науч. 2023. PMID: 36959928 Бесплатная статья ЧВК.
- Включение ковариатов, полученных из почвы, в тестирование потомства и отбор линий для повышения точности геномного прогнозирования в селекции сои.
Канелла Виейра С., Перса Р., Чен П., Харкин Д. Канелла Виейра С. и др. Фронт Жене. 2022, 8 сентября; 13:
- 4. дои: 10.3389/fgene.2022.
- 4.
 Электронная коллекция 2022.
Фронт Жене. 2022.
PMID: 36159995
Бесплатная статья ЧВК.
Электронная коллекция 2022.
Фронт Жене. 2022.
PMID: 36159995
Бесплатная статья ЧВК.
Просмотреть все статьи «Цитируется по»
Рекомендации
- Ниблэк Т.Л., Ламберт К.Н., Тылка Г.Л. Модельный патоген растений из царства животных: Heterodera glycines, цистообразующая нематода сои. Анну Рев Фитопат. 2006; 44: 283–303. doi: 10.1146/annurev.phyto.43.040204.140218. — DOI — пабмед
- Кеннинг С.Р., Вратер Дж.А. Подавление потенциальной урожайности сои в континентальной части США из-за болезней растений с 2006 по 2009 год.
 Прогресс в области здоровья растений. 2010. doi:10.1094/PHP-2010-1122-01-RS.
Прогресс в области здоровья растений. 2010. doi:10.1094/PHP-2010-1122-01-RS.
- Кеннинг С.Р., Вратер Дж.А. Подавление потенциальной урожайности сои в континентальной части США из-за болезней растений с 2006 по 2009 год.
- Concibido VC, Diers BW, Arelli PR. Десятилетие картирования QTL устойчивости к цистообразным нематодам у сои. Растениеводство. 2004;44(4):1121–31. doi: 10.2135/cropsci2004.1121. — DOI
- Феррейра МФД, Червиньи ГДЛ, Феррейра А., Шустер И., Сантана Ф.А., Перейра В.Д. и др. QTL для устойчивости к соевой нематоде, расы 3, 9 и 14 у сорта Хартвиг. Бюстгальтеры Pesqui Agropecu. 2011;46(4):420–8. doi: 10.1590/S0100-204X2011000400012. — DOI
- Го Б.
 , Слепер Д.А., Нгуен Х.Т., Арелли П.Р., Шеннон Дж.Г. Локусы количественных признаков, лежащие в основе устойчивости к трем популяциям цистообразующих нематод сои в сое PI 404198A. Растениеводство. 2006;46(1):224–33. doi: 10.2135/cropsci2004.0757.
—
DOI
, Слепер Д.А., Нгуен Х.Т., Арелли П.Р., Шеннон Дж.Г. Локусы количественных признаков, лежащие в основе устойчивости к трем популяциям цистообразующих нематод сои в сое PI 404198A. Растениеводство. 2006;46(1):224–33. doi: 10.2135/cropsci2004.0757.
—
DOI
- Го Б.
Типы публикаций
термины MeSH
вещества
Однонуклеотидный полиморфизм — ISOGG Wiki
Другие языки:
Английский
Однонуклеотидный полиморфизм ( SNP , произносится как snip ) представляет собой вариацию последовательности ДНК, возникающую, когда один нуклеотид аденин (A), тимин (T), цитозин (C) или гуанин (G]) в геноме (или другой общей последовательности) отличается между представителями вида или парными хромосомами у человека. Например, два секвенированных фрагмента ДНК разных людей, от AAGC C TA до AAGC T TA, содержат различие в одном нуклеотиде. В этом случае мы говорим, что есть два аллели : C и T. Почти все распространенные SNP имеют только два аллеля.
Например, два секвенированных фрагмента ДНК разных людей, от AAGC C TA до AAGC T TA, содержат различие в одном нуклеотиде. В этом случае мы говорим, что есть два аллели : C и T. Почти все распространенные SNP имеют только два аллеля.
Внутри популяции SNP может быть присвоена частота минорного аллеля — самая низкая частота аллеля в локусе, наблюдаемая в конкретной популяции. Это просто меньшая из двух частот аллелей для однонуклеотидных полиморфизмов. Существуют различия между человеческими популяциями, поэтому аллель SNP, распространенный в одной географической или этнической группе, может быть гораздо реже в другой.
Содержание
- 1 Типы SNP
- 2 Список SNP
- 3 Использование и важность SNP
- 4 Дальнейшее чтение
- 5 ресурсов
- 6 баз данных
- 7 Полезные инструменты для опытных пользователей
- 8 См. также
Типы SNP
Отдельные нуклеотиды могут быть изменены (замена), удалены (делеции) или добавлены (вставка) в полинуклеотидную последовательность.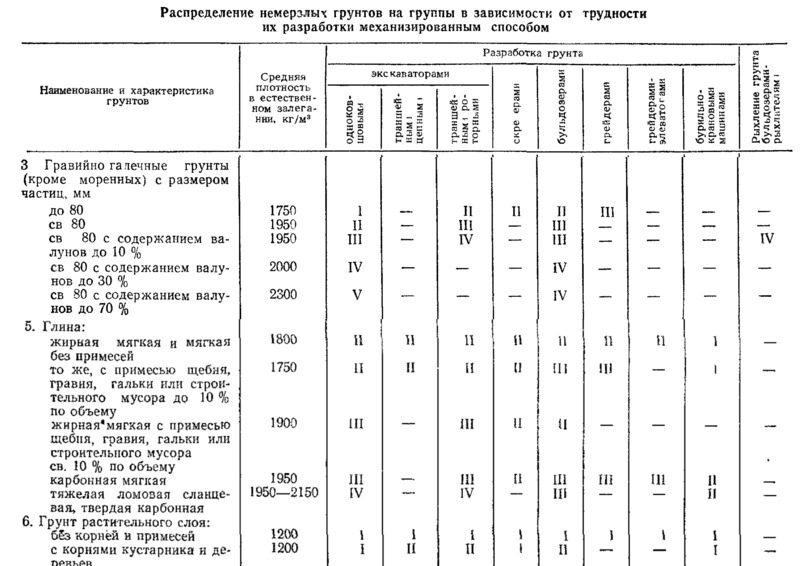 Однонуклеотидные полиморфизмы могут находиться в кодирующих последовательностях генов, некодирующих областях генов или в межгенных областях между генами. SNP в кодирующей последовательности не обязательно изменят аминокислотную последовательность продуцируемого белка из-за вырождения генетического кода.
Однонуклеотидные полиморфизмы могут находиться в кодирующих последовательностях генов, некодирующих областях генов или в межгенных областях между генами. SNP в кодирующей последовательности не обязательно изменят аминокислотную последовательность продуцируемого белка из-за вырождения генетического кода.
SNP, в котором обе формы приводят к одной и той же полипептидной последовательности, называется синонимичной (иногда называемой молчащей мутацией) — если продуцируется другая полипептидная последовательность, они несинонимичны . Несинонимическое изменение может быть либо миссенс, либо нонсенсом, где миссенс-изменение приводит к другой аминокислоте, а бессмысленное изменение приводит к преждевременному стоп-кодону. SNP, которые не находятся в областях, кодирующих белок, могут по-прежнему иметь последствия для сплайсинга генов, связывания фактора транскрипции или последовательности некодирующей рибонуклеиновой кислоты (РНК).
Список SNP
Подтвержденный SNP Z282 (зеленый цвет означает положительный результат + или подтвержденный тестированием SNP) из FTDNA. На этом снимке экрана показаны различные цвета, используемые для обозначения SNP.
На этом снимке экрана показаны различные цвета, используемые для обозначения SNP.SNP, в частности SNP ключевого маркера, используемого в гапологруппах, обозначаются цветом или кодом в генетической генеалогии.
Непроверенный или оцененный SNP обычно обозначается черным или стандартным цветом текста или без символа проверенного кода подтверждения (+) или неподтверждения (-).
Протестированный SNP, подтвержденный тестированием SNP, обычно обозначается текстом зеленого цвета или знаком плюса (+) после обозначения SNP. Его часто называют , производным от , или ему присваивают положительный (+) код.
Протестированный SNP, который не подтвержден или является отрицательным (-), обычно выделяется красным цветом.
SNP, ожидающие тестирования или предположительно отрицательные, имеют разные цвета, но не используют код ни положительного (+), ни отрицательного (-). Разные тестовые компании могут использовать разные цвета, поэтому важно подтвердить обозначения в приведенных таблицах.
Для примера гаплогруппы R1a1a1b1a с маркером SNP Z282 и использованием сокращенного кода R-Z282 используются назначенные цвета, представляющие статус SNP. Смотрите изображение.
Использование и значение SNP
Изменения в последовательностях ДНК человека могут влиять на то, как у людей развиваются заболевания и как они реагируют на патогены, химические вещества, лекарства, вакцины и другие агенты. SNP также считаются ключевыми факторами реализации концепции персонализированной медицины. Однако их наибольшее значение в биомедицинских исследованиях заключается в сравнении областей генома между когортами (например, совпадающими когортами с заболеванием и без него).
Дополнительная литература
- Азбука ДНК — SNP Касандры Роуз, Следы ДНК , 24 декабря 2016 г.
- Мастер-класс DNA Heritage по SNP и гаплогруппам (ссылка на Интернет-архив)
Ресурсы
- Индекс ISOGG SNP
- Информация о проекте «Геном человека» — информационный бюллетень SNP Ресурсы
- NCBI — Введение в SNP из NCBI
- ООО «Консорциум СНП» — Поиск СНП
- SNPedia — вики, посвященная медицинским последствиям вариаций ДНК, включая программное обеспечение для анализа личных геномов
- Руководство HGNC по номенклатуре генов человека
- Сборник SNP Дэвида Рейнольдса
- Базы данных генетических вариаций — ресурс Центра клинической генетики человека со ссылками на веб-сайты для: баз данных SNP, программного обеспечения для разработки праймеров, специфичных для SNP; обнаружение SNP и предсказание эффекта; и болезнетворные вариации.

Базы данных
- Проект «1000 геномов» — подробный каталог генетической изменчивости человека
- База данных NCBI dbSNP — «Центральный репозиторий как одноосновных нуклеотидных замен, так и полиморфизмов коротких делеций и вставок»
- GWAS Central — GWAS Central (ранее известная как База данных вариаций генома человека, содержащая информацию о генотипе и фенотипе) — это центральное хранилище результатов полногеномных ассоциативных исследований (GWAS). Он включает информацию обо всех известных SNP и других вариантах из общедоступных баз данных, таких как dbSNP и DBGV.
- PharmGKB — База знаний по фармакогенетике и фармакогеномике, ресурс для SNP, связанных с ответом на лекарство и исходами заболевания
- Международный проект HapMap — «Общественный ресурс, который поможет исследователям найти гены, связанные с болезнями человека и реакцией на фармацевтические препараты»; обратите внимание, что проект HapMap был прекращен в июне 2016 г.

 0,25
0,25 1,0
1,0