Раскружаливание конструкций стадионов к чемпионатам мира 2018 г.
Продукция
Каталоги
-
Проекты Мостмеханика
-
Гидравлический инструмент
-
Силовой такелаж
-
Гидравлическое оборудование для строительной отрасли
-
Бескрановые технологии. Скиддинг системы СКАТ
Новости
2023/04/14 «НОВАТЭК» провел взвешивание первого СПГ-модуля синхронизированной гидравлической системой «Мостмеханика»
В центре строительства крупнотоннажных морских сооружений в Белокаменке «НОВАТЭК» провел взвешивание первого СПГ-модуля синхронизированной гидравлической системой «Мостмеханика» .
Еще новости…
Главная страница→Новости→Раскружаливание конструкций стадионов к чемпионатам мира 2018 г.
24.10.2019
Компания «Мостмеханика» принимала участие в строительстве стадионов «Екатеринбург Арена» в г. Екатеринбурге, «Самара Арена» в г. Самаре, «Мордовия Арена» в г. Саранске к чемпионату мира 2018 года. Благодаря системам многоточечного синхронного подъема конструкций кровель стадионов СМР, разработанным компанией «Мостмеханика», в кратчайшие сроки были выполнены работы по раскружаливанию. Работы выполнялись специалистами «Мостмеханика» на условиях субподряда.
Раскружаливание – важнейший этап строительства стадиона, включающий в себя комплекс работ по замещению смонтированных конструкций купола путем постепенного извлечения временных опор. Все работы проходят под контролем специалистов осуществляющих строительный и технологический надзор, а также под геодезическим контролем положения металлоконструкций.
Фактическое время раскружаливания: подъем кровли и опускание на постоянные опоры составлял в среднем 40 часов.
Системы многоточечного подъема и раскружаливания кровли строительных объектов комплектуются индивидуально под проект Заказчика. К примеру типовая комплектация СМР состоит из:
- 204-х специальных гидравлических домкратов и гидроцилиндров;
- 4-х маслостанций;
- 88 блоков управления, комплекта гидроэлектроарматуры;
- 3150 метров рукавов высокого давления.
Предлагаем Вашему вниманию буклет со значимыми объектами при возведении которых применялись технологии и оборудование «Мостмеханика».
| «Екатеринбург Арена» в г. Екатеринбурге | ||
«Мордовия Арена» в г. Саранск Саранск |
||
| «Самара Арена» в г. Самара | ||
« вернуться
43. Механизмы для раскружаливания арочных пролетных строений.
Раскружаливание арок – это процесс плавного введения их в работу путем постепенной разгрузки кружал. Приступать к этому процессу можно после достижения бетоном клиньев 100% прочности.
До раскружаливания необходимо:
1.определить в бетонной лаборатории фактическую прочность бетона арок с помощью испытаний контрольных образцов;
2.снять опалубку с боковых поверхностей и визуально освидетельствовать состояние бетона арок.
Осаживание
(опускание) стоечных и башенных кружал
производится последовательно по
направлению от середины пролета к
опорам. Наибольшая величина перемещения
арки в местах размещения приборов
раскружаливания определяется с учетом
упругого обжатия арки и упругой деформации
кружал. Это связано с тем, что при снятии
нагрузки кружала, освобождаясь, как бы
приподнимаются вверх, а затем происходит
упругое обжатие бетона арки, то есть
приборы раскружаливания работают, а
зазора между аркой и кружалами не
появляется.
Наибольшая величина перемещения
арки в местах размещения приборов
раскружаливания определяется с учетом
упругого обжатия арки и упругой деформации
кружал. Это связано с тем, что при снятии
нагрузки кружала, освобождаясь, как бы
приподнимаются вверх, а затем происходит
упругое обжатие бетона арки, то есть
приборы раскружаливания работают, а
зазора между аркой и кружалами не
появляется.
В качестве приборов раскружаливания чаще всего используют деревянные клинья (при малых пролетах), песочницы и гидравлические домкраты.
Раскружаливание забетонированной арки (свода) на арочных кружалах производится в пятах или в замке после набора бетоном необходимой прочности.
Рис, 4.27 – Способы раскружаливания арок: а – опускание в пятах; б – опускание в замке; в – поднятие свода домкратами
При
первом способе (рис. 4.27, а), когда приборы
раскружаливания размещаются в пятах,
распор сохраняется до конца опускания.
Это создает определенные неудобства,
поскольку кружала зажаты и в них действуют
усилия, что требуется учитывать при
демонтаже кружал.
Второй способ (опускание кружал в замке) более эффективен. Песочницы устанавливают горизонтально в замке (рис. 4.27, б), и распор постепенно передается на арки. Способ предпочтителен для трехшарнирной арки пролетом до 60 м.
Третий способ – раскружаливание домкратами, установленными в замке арки (рис. 4.27, в). При работе домкратов арка плавно снимается с кружал. После этого на нее передается усилие от распора, а домкраты освобождаются. В арке, таким образом, создаются начальные усилия, обратные по знаку усилиям, возникающим от упругого обжатия, ползучести и усадки бетона. Вследствие этого удается избежать провисания арки в последующем.
Временная опора чаще всего выполняется в виде пространственной металлической конструкции башенного типа из элементов МИК–С (рис. 6.37), а также верхнего и нижнего сварных ростверков из металлических балок.
Рис. 6.37 – Временная опора при полунавесном монтаже: а – нагрузки на опору; б – фасад; в – схемы к расчету оголовка; 1 – сборочная клетка; 2 – продольные балки; 3 – поперечная балка
На верхний ростверк
укладывают металлические балки, на
которых размещают сборочные клетки и
домкраты (рис.
Рис. 6.38 – Узел опирания пролетного строения на временную опору при полунавесном монтаже: 1 – гидравлические домкраты, объединенные в батарею; 2 – металлические пакеты и клетки
Через нижние ростверки опора опирается на металлические, деревянные или железобетонные сваи или иное жесткое основание. Пример такой опоры из элементов МИК–С для монтажа автодорожного пролетного строения приведен на (рис. 6.39).
Рис. 6.39 – Пример конструкции временной опоры из элементов МИК–С: 1–11 – марки элементов
Структура эукариотической ДНК-полимеразы δ, связанной с зажимом PCNA при окружении ДНК
1. Гулбис Дж. М., Келман З., Гурвиц Дж., О’Доннелл М., Куриян Дж., Структура С-концевой области р21 ( WAF1/CIP1) в комплексе с PCNA человека. Клетка 87, 297–306 (1996). [PubMed] [Google Scholar]
2. Kong X.P., Onrust R., O’Donnell M., Kuriyan J., Трехмерная структура бета-субъединицы холофермента ДНК-полимеразы III E. coli: скользящий ДНК-зажим.
3. Кришна Т.С., Конг Х.П., Гэри С., Бургерс П.М., Куриян Дж., Кристаллическая структура фактора процессивности эукариотической ДНК-полимеразы PCNA. Клетка 79, 1233–1243 (1994). [PubMed] [Google Scholar]
4. Куриян Дж., О’Доннелл М., Скользящие зажимы ДНК-полимераз. Дж. Мол. биол. 234, 915–925 (1993). [PubMed] [Google Scholar]
5. Stukenberg P.T., Studwell-Vaughan P.S., O’Donnell M., Механизм скользящего бета-зажима холофермента ДНК-полимеразы III. Дж. Биол. хим. 266, 11328–11334 (1991). [PubMed] [Google Scholar]
6. Джерузалми Д., О’Доннелл М., Куриян Дж., Загрузчики с зажимами и скользящие зажимы. Курс. мнение Структура биол. 12, 217–224 (2002). [PubMed] [Google Scholar]
7. Яо Н., О’Доннелл М., Бактериальные и эукариотические реплисомные машины. JSM Биохим. Мол. биол. 3, 1013 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
8. Брейтуэйт Д. К., Ито Дж., Компиляция, выравнивание и филогенетические взаимоотношения ДНК-полимераз. Нуклеиновые Кислоты Res.
21, 787–802 (1993). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Нуклеиновые Кислоты Res.
21, 787–802 (1993). [Бесплатная статья PMC] [PubMed] [Google Scholar]
9. Burgers P.M.J., Kunkel T.A., Вилка репликации эукариотической ДНК. Анну. Преподобный Биохим. 86, 417–438 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
10. Кункель Т. А., Бюргерс П. М., Разделение рабочей нагрузки на эукариотической вилке репликации. Тенденции клеточной биологии. 18, 521–527 (2008). [Статья бесплатно PMC] [PubMed] [Google Scholar]
11. Джорджеску Р. Э. и др., Механизм сборки асимметричной полимеразы на эукариотической репликационной вилке. Нац. Структура Мол. биол. 21, 664–670 (2014). [Бесплатная статья PMC] [PubMed] [Google Scholar]
12. Langston L.D., et al., Хеликаза CMG и ДНК-полимераза ε образуют функциональный 15-субъединичный холофермент для репликации ведущей цепи эукариотической ДНК. проц. Натл. акад. науч. США. 111, 15390–15395 (2014). [Бесплатная статья PMC] [PubMed] [Google Scholar]
13. Ария В., Йилес Дж. Т. П., Механизм установления двунаправленного синтеза ведущей цепи в местах начала репликации эукариотической ДНК. Мол. Клетка
73, 199–211 (2018). [Статья бесплатно PMC] [PubMed] [Google Scholar]
П., Механизм установления двунаправленного синтеза ведущей цепи в местах начала репликации эукариотической ДНК. Мол. Клетка
73, 199–211 (2018). [Статья бесплатно PMC] [PubMed] [Google Scholar]
14. Чжоу З. Х., Лужан С. А., Буркхолдер А. Б., Гарбач М. А., Кункель Т. А., Роль ДНК-полимеразы δ в инициации и терминации репликации ДНК ведущей цепи. Нац. коммун. 10, 3992 (2019). [Бесплатная статья PMC] [PubMed] [Google Scholar]
15. Джорджеску Р., Лэнгстон Л., О’Доннелл М. Предложение: эволюция роли PCNA в качестве маркера недавно реплицированной ДНК. Восстановление ДНК (Amst.) 29, 4–15 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
16. Стодола Дж. Л., Бургерс П. М., Разрешение отдельных этапов созревания фрагментов Окадзаки в миллисекундном масштабе. Нац. Структура Мол. биол. 23, 402–408 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
17. Альтьери А. С., Кельман З., Скользящие зажимы ДНК как терапевтические мишени. Передний. Мол. Бионауч.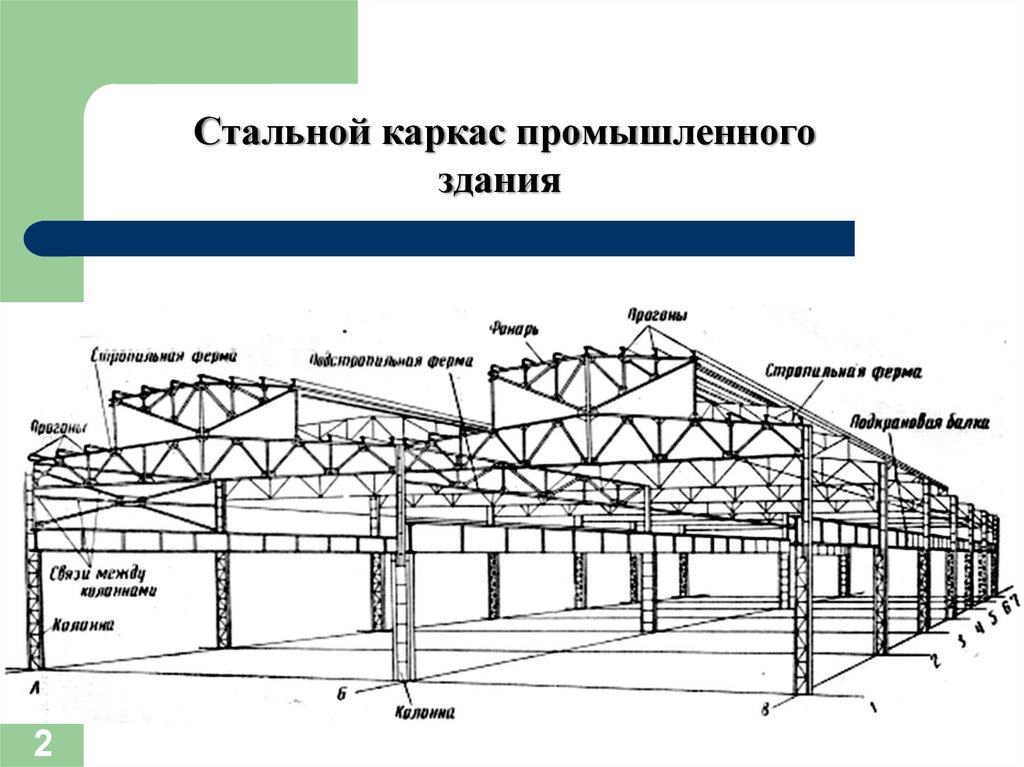 5, 87 (2018). [Бесплатная статья PMC] [PubMed] [Google Scholar]
5, 87 (2018). [Бесплатная статья PMC] [PubMed] [Google Scholar]
18. Dalrymple B.P., Kongsuwan K., Wijffels G., Dixon N.E., Jennings P.A., Универсальный мотив белок-белкового взаимодействия в системах репликации и восстановления ДНК эубактерий. проц. Натл. акад. науч. США. 98, 11627–11632 (2001). [Бесплатная статья PMC] [PubMed] [Google Scholar]
19. Boehm E.M., Washington M.T., R.I.P. к PIP: мотив связывания PCNA больше не считается специфичным: мотивы PIP и другие родственные последовательности не являются отдельными объектами и могут связывать несколько белков, участвующих в поддержании генома. Биоэссе 38, 1117–1122 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
20. Netz D.J., et al., Эукариотические ДНК-полимеразы нуждаются в железо-серном кластере для образования активных комплексов. Нац. хим. биол. 8, 125–132 (2011). [Статья бесплатно PMC] [PubMed] [Google Scholar]
21. Лэнгстон Л. Д., О’Доннелл М., ДНК-полимераза дельта высоко процессивна с ядерным антигеном пролиферирующих клеток и подвергается высвобождению при столкновении после завершения ДНК. Дж. Биол. хим.
283, 29522–29531 (2008). [Статья бесплатно PMC] [PubMed] [Google Scholar]
Дж. Биол. хим.
283, 29522–29531 (2008). [Статья бесплатно PMC] [PubMed] [Google Scholar]
22. O’Donnell M. E. Дополнительные белки связывают праймированную матрицу и опосредуют быстрый цикл холофермента ДНК-полимеразы III из Escherichia coli. Дж. Биол. хим. 262, 16558–16565 (1987). [PubMed] [Google Scholar]
23. Стукенберг П. Т., Тернер Дж., О’Доннелл М., Объяснение репликации отстающих цепей: прыжки полимеразы между скользящими зажимами ДНК. Клетка 78, 877–887 (1994). [PubMed] [Google Scholar]
24. Битти Т. Р., Белл С. Д., Координация активности нескольких ферментов с помощью одного PCNA при созревании фрагментов архей Окадзаки. ЭМБО Дж. 31, 1556–1567 (2012). [Бесплатная статья PMC] [PubMed] [Google Scholar]
25. Кельман Л. М., Кельман З., Репликация ДНК архей. Анну. Преподобный Жене. 48, 71–97 (2014). [PubMed] [Google Scholar]
26. Guilliam T.A., Yeeles JTP. Реконструкция синтеза трансфекции раскрывает механизм перезапуска репликации эукариотической ДНК. Нац. Структура Мол. биол.
27, 450–460 (2020). [Статья бесплатно PMC] [PubMed] [Google Scholar]
Нац. Структура Мол. биол.
27, 450–460 (2020). [Статья бесплатно PMC] [PubMed] [Google Scholar]
27. Доннианни Р. А. и др. Дельта-ДНК-полимераза синтезирует обе цепи во время репликации, индуцированной разрывом. Мол. Клетка 76, 371–381.e4 (2019). [Статья бесплатно PMC] [PubMed] [Google Scholar]
28. Huang J., et al., Формирование интерференционно-чувствительных мейотических кроссинговеров требует достаточного удлинения ведущей цепи ДНК. проц. Натл. акад. науч. США. 112, 12534–12539(2015). [PMC free article] [PubMed] [Google Scholar]
29. Johnson R.E., Prakash L., Prakash S., Pol31 и Pol32 субъединицы дрожжевой ДНК-полимеразы δ также являются важными субъединицами ДНК-полимеразы ζ. проц. Натл. акад. науч. США. 109, 12455–12460 (2012). [Статья бесплатно PMC] [PubMed] [Google Scholar]
30. Джайн Р. и др., Крио-ЭМ структура и динамика эукариотической ДНК-полимеразы δ холофермента. Нац. Структура Мол. биол. 26, 955–962 (2019). [Бесплатная статья PMC] [PubMed] [Google Scholar]
31. Lancey C., et al., Структура процессивного холофермента Pol δ человека. Нац. коммун.
11, 1109 (2020). [Статья бесплатно PMC] [PubMed] [Google Scholar]
Lancey C., et al., Структура процессивного холофермента Pol δ человека. Нац. коммун.
11, 1109 (2020). [Статья бесплатно PMC] [PubMed] [Google Scholar]
32. Madru C., et al., Структурная основа повышенной процессивности ДНК-полимераз D-семейства в комплексе с PCNA. Нац. коммун. 11, 1591 (2020). [Статья бесплатно PMC] [PubMed] [Google Scholar]
33. Fernandez-Leiro R., Conrad J., Scheres S.H., Lamers M.H., крио-ЭМ структуры репликативной ДНК-полимеразы E. coli показывают ее динамическое взаимодействие с Скользящий зажим ДНК, экзонуклеаза и τ. электронная жизнь 4, e11134 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
34. Ачарья Н., Классен Р., Джонсон Р. Э., Пракаш Л., Пракаш С., PCNA-связывающие домены во всех трех субъединицах дрожжевой ДНК-полимеразы δ модулируют ее функцию в репликации ДНК. проц. Натл. акад. науч. США. 108, 17927–17932 (2011). [Статья бесплатно PMC] [PubMed] [Google Scholar]
35. Barros T., et al., Структурная роль домена PHP в ДНК-полимеразе III E. coli. Структура БМК. биол.
13, 8 (2013). [Статья бесплатно PMC] [PubMed] [Google Scholar]
coli. Структура БМК. биол.
13, 8 (2013). [Статья бесплатно PMC] [PubMed] [Google Scholar]
36. Hedglin M., Pandey B., Benkovic S.J., Стабильность холофермента полимеразы δ человека и его значение в синтезе отстающей цепи ДНК. проц. Натл. акад. науч. США. 113, E1777–E1786 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
37. Кэмпбелл Б. Б. и др. Всесторонний анализ гипермутаций при раке человека. Клетка 171, 1042–1056.e10 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
38. да Коста Л. Т. и др., Дельта-варианты полимеразы в колоректальных опухолях RER. Нац. Жене. 9, 10–11 (1995). [PubMed] [Google Scholar]
39. Elouej S., et al., Секвенирование экзома выявило мутацию de novo POLD1, вызывающую фенотипическую изменчивость гипоплазии нижней челюсти, глухоты, прогероидных признаков и синдрома липодистрофии (MDPL). Метаболизм 71, 213–225 (2017). [PubMed] [Академия Google]
40. Flohr T., et al., Обнаружение мутаций в гене ДНК-полимеразы дельта спорадического колоректального рака человека и клеточных линий рака толстой кишки. Междунар. Дж. Рак
80, 919–929 (1999). [PubMed] [Google Scholar]
Междунар. Дж. Рак
80, 919–929 (1999). [PubMed] [Google Scholar]
41. Rayner E., et al., Набор ошибок: мутации корректирующего домена полимеразы при раке. Нац. Преподобный Рак 16, 71–81 (2016). [PubMed] [Google Scholar]
42. Джорджеску Р. Э. и др. Структура скользящего зажима на ДНК. Клетка 132, 43–54 (2008). [Бесплатная статья PMC] [PubMed] [Google Scholar]
43. McNally R., Bowman G.D., Goedken E.R., O’Donnell M., Kuriyan J., Анализ роли контактов PCNA-ДНК во время нагрузки зажима. Структура БМК. биол. 10, 3 (2010). [Статья бесплатно PMC] [PubMed] [Google Scholar]
44. Klinge S., Hirst J., Maman J.D., Krude T., Pellegrini L., Железо-серный домен эукариотической примазы необходим для синтеза РНК-праймера. . Нац. Структура Мол. биол. 14, 875–877 (2007). [Статья бесплатно PMC] [PubMed] [Google Scholar]
45. Джорджеску Р. Э. и др., Структура низкомолекулярного ингибитора скользящего зажима ДНК-полимеразы. проц. Натл. акад. науч. США.
105, 11116–11121 (2008).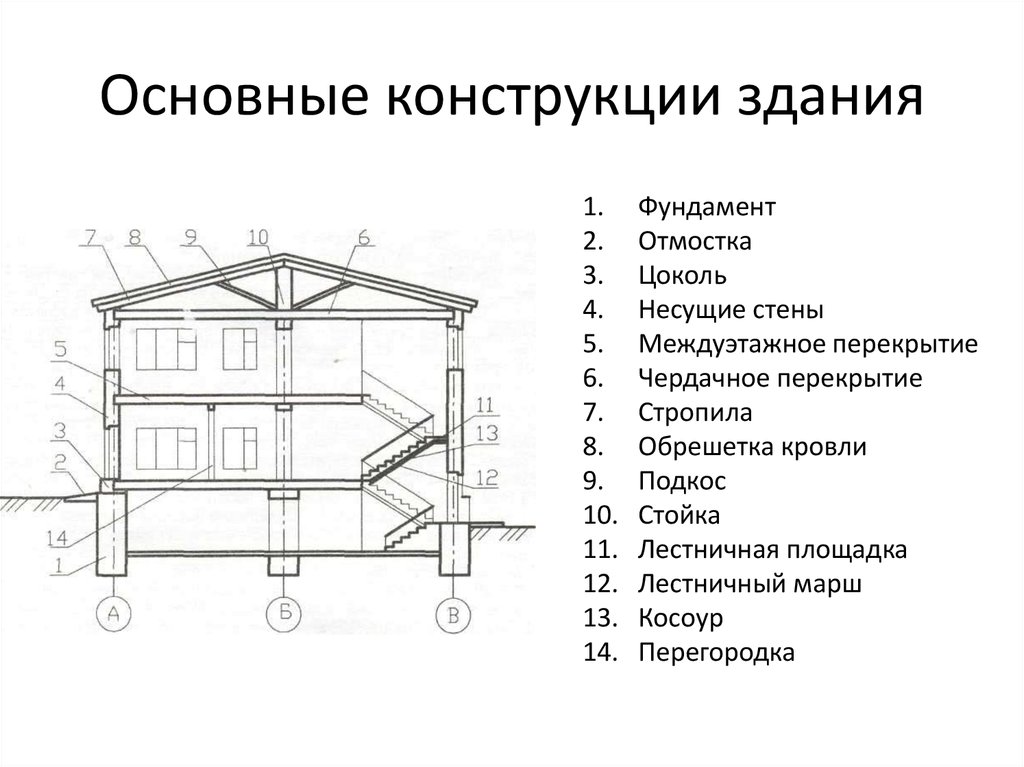 [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]
46. Huang M.E., Le Douarin B., Henry C., Galibert F., Белок Saccharomyces cerevisiae YJR043C (Pol32) взаимодействует с каталитической субъединицей ДНК-полимеразы альфа и необходим для прогрессирования клеточного цикла в G2/M. Мол. Генерал Жене. 260, 541–550 (1999). [PubMed] [Google Scholar]
47. Johansson E., Garg P., Burgers PM, Субъединица Pol32 ДНК-полимеразы дельта содержит разделяемые домены для процессивной репликации и связывания ядерного антигена пролиферирующих клеток (PCNA). Дж. Биол. хим. 279, 1907–1915 (2004). [PubMed] [Google Scholar]
48. Льюис Дж. С. и др., Настройка стабильности ДНК-полимеразы во время репликации эукариотической ДНК. Мол. Клетка 77, 17–25.e5 (2020). [Бесплатная статья PMC] [PubMed] [Google Scholar]
49. Yuan Z., et al., Ctf4 организует сестринские реплисомы и Pol α в фабрику репликации. электронная жизнь 8, e47405 (2019). [Бесплатная статья PMC] [PubMed] [Google Scholar]
50. Yao N.Y., Georgescu R.E., Finkelstein J., O’Donnell M.E., Анализ одиночных молекул показывает, что отстающая цепь увеличивает процессивность реплисом, но замедляет прогрессирование репликативной вилки. проц. Натл. акад. науч. США.
106, 13236–13241 (2009 г.)). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Yao N.Y., Georgescu R.E., Finkelstein J., O’Donnell M.E., Анализ одиночных молекул показывает, что отстающая цепь увеличивает процессивность реплисом, но замедляет прогрессирование репликативной вилки. проц. Натл. акад. науч. США.
106, 13236–13241 (2009 г.)). [Бесплатная статья PMC] [PubMed] [Google Scholar]
51. Индиани С., Макинерни П., Джорджеску Р., Гудман М. Ф., О’Доннелл М., Ремень со скользящим зажимом связывает ДНК с высокой и низкой точностью. полимеразы одновременно. Мол. Клетка 19, 805–815 (2005). [PubMed] [Google Scholar]
52. Стодола Дж. Л., Бюргерс П. М., Механизм репликации ДНК с запаздывающей цепью у эукариот. Доп. Эксп. Мед. биол. 1042, 117–133 (2017). [PubMed] [Google Scholar]
53. Дионн И., Ноокала Р.К., Джексон С.П., Доэрти А.Дж., Белл С.Д., Гетеротримерный PCNA в гипертермофильных археях Sulfolobus solfataricus. Мол. Клетка 11, 275–282 (2003). [PubMed] [Академия Google]
54. Mayanagi K., et al., Механизм сборки механизма репликации, выявленный архитектурой комплекса ДНК-лигаза-PCNA-ДНК. проц. Натл. акад. науч. США.
106, 4647–4652 (2009). [Бесплатная статья PMC] [PubMed] [Google Scholar]
проц. Натл. акад. науч. США.
106, 4647–4652 (2009). [Бесплатная статья PMC] [PubMed] [Google Scholar]
55. Доврат Д., Стодола Дж. Л., Бургерс П. М., Ахарони А., Последовательное переключение партнеров по связыванию на PCNA во время созревания фрагмента Окадзаки in vitro. проц. Натл. акад. науч. США. 111, 14118–14123 (2014). [Бесплатная статья PMC] [PubMed] [Google Scholar]
56. Чжэн С. К. и др., MotionCor2: Анизотропная коррекция движения, вызванного лучом, для улучшения криоэлектронной микроскопии. Нац. Методы 14, 331–332 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
57. Рохоу А., Григорьев Н., CTFFIND4: Быстрая и точная оценка расфокусировки по электронным микрофотографиям. Дж. Структура. биол. 192, 216–221 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
58. Зиванов Дж. и др. Новые инструменты для автоматизированного определения структуры крио-ЭМ высокого разрешения в РЕЛИОН-3. электронная жизнь 7, e42166 (2018). [Бесплатная статья PMC] [PubMed] [Google Scholar]
59. Пунджани А., Рубинштейн Дж. Л., Флит Д. Дж., Брубейкер М. А., cryoSPARC: Алгоритмы для быстрого неконтролируемого определения структуры крио-ЭМ. Нац. Методы
14. С. 290–296 (2017). [PubMed] [Google Scholar]
Пунджани А., Рубинштейн Дж. Л., Флит Д. Дж., Брубейкер М. А., cryoSPARC: Алгоритмы для быстрого неконтролируемого определения структуры крио-ЭМ. Нац. Методы
14. С. 290–296 (2017). [PubMed] [Google Scholar]
60. Адамс П. Д. и др., PHENIX: комплексная система на основе Python для решения макромолекулярной структуры. Акта Кристаллогр. Д биол. Кристаллогр. 66, 213–221 (2010). [Бесплатная статья PMC] [PubMed] [Google Scholar]
61. Emsley P., Lohkamp B., Scott W.G., Cowtan K., Особенности и разработка Coot. Акта Кристаллогр. Д биол. Кристаллогр. 66, 486–501 (2010). [Бесплатная статья PMC] [PubMed] [Google Scholar]
62. Chen V.B., et al., MolProbity: Проверка полноатомной структуры для макромолекулярной кристаллографии. Акта Кристаллогр. Д биол. Кристаллогр. 66, 12–21 (2010). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Структурная архитектура длинной некодирующей РНК человека, активатор РНК стероидного рецептора
1. Okazaki Y, Furuno M, Kasukawa T, Adachi J, Bono H, Kondo S , Никайдо И. , Осато Н., Сайто Р., Судзуки Х. и др. Анализ транскриптома мыши на основе функциональной аннотации 60 770 полноразмерных кДНК. Природа. 2002; 420: 563–573. [PubMed] [Академия Google]
, Осато Н., Сайто Р., Судзуки Х. и др. Анализ транскриптома мыши на основе функциональной аннотации 60 770 полноразмерных кДНК. Природа. 2002; 420: 563–573. [PubMed] [Академия Google]
2. Ван Дж., Чжан Дж., Чжэн Х., Ли Дж., Лю Д., Ли Х., Самудрала Р., Ю Дж., Вонг Г.К. Транскриптом мыши: нейтральная эволюция «некодирующих» комплементарных ДНК. Природа. 2004;431 1 п. после 757; обсуждение после 757. [PubMed] [Google Scholar]
3. Carthew RW, Sontheimer EJ. Происхождение и механизмы miRNAs и siRNAs. Клетка. 2009; 136: 642–655. [Бесплатная статья PMC] [PubMed] [Google Scholar]
4. Mercer TR, Dinger ME, Mattick JS. Длинные некодирующие РНК: понимание функций. Нац. Преподобный Жене. 2009 г.;10:155–159. [PubMed] [Google Scholar]
5. Mercer TR, Dinger ME, Sunkin SM, Mehler MF, Mattick JS. Специфическая экспрессия длинных некодирующих РНК в мозге мыши. проц. Натл акад. науч. США. 2008; 105: 716–721. [Бесплатная статья PMC] [PubMed] [Google Scholar]
6. Wapinski O, Chang HY. Длинные некодирующие РНК и болезни человека. Тенденции клеточной биологии. 2011;21:354–361. [PubMed] [Google Scholar]
Длинные некодирующие РНК и болезни человека. Тенденции клеточной биологии. 2011;21:354–361. [PubMed] [Google Scholar]
7. Wang X, Song X, Glass CK, Rosenfeld MG. Длинное плечо длинных некодирующих РНК: роль сенсоров, регулирующих программы транскрипции генов. Харб Колд Спринг. Перспектива. биол. 2011;3:a003756. [Бесплатная статья PMC] [PubMed] [Google Scholar]
8. Dinger ME, Amaral PP, Mercer TR, Pang KC, Bruce SJ, Gardiner BB, Askarian-Amiri ME, Ru K, Soldà G, Simons C, et al. Длинные некодирующие РНК в плюрипотентности и дифференцировке эмбриональных стволовых клеток мыши. Геном Res. 2008; 18:1433–1445. [Бесплатная статья PMC] [PubMed] [Google Scholar]
9. Саттерли Дж. С., Барби С., Джин П., Кричевский А., Салама С., Шратт Г., Ву Д.Ю. Некодирующие РНК в головном мозге. Дж. Нейроски. 2007; 27:11856–11859. [Бесплатная статья PMC] [PubMed] [Google Scholar]
10. Kaikkonen MU, Lam MT, Glass CK. Некодирующие РНК как регуляторы экспрессии генов и эпигенетика. Кардиовас. Рез. 2011;90: 430–440. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Кардиовас. Рез. 2011;90: 430–440. [Бесплатная статья PMC] [PubMed] [Google Scholar]
11. Tsai MC, Manor O, Wan Y, Mosammaparast N, Wang JK, Lan F, Shi Y, Segal E, Chang HY. Длинная некодирующая РНК как модульный каркас комплексов модификации гистонов. Наука. 2010; 329: 689–693. [Бесплатная статья PMC] [PubMed] [Google Scholar]
12. Tian D, Sun S, Lee JT. Длинная некодирующая РНК, jpx, представляет собой молекулярный переключатель для инактивации Х-хромосомы. Клетка. 2010; 143:390–403. [Бесплатная статья PMC] [PubMed] [Google Scholar]
13. Heo JB, Sung S. Опосредованное яровизацией эпигенетическое молчание с помощью длинной интронной некодирующей РНК. Наука. 2011; 331:76–79. [PubMed] [Google Scholar]
14. Wang KC, Yang YW, Liu B, Sanyal A, Corces-Zimmerman R, Chen Y, Lajoie BR, Protacio A, Flynn RA, Gupta RA, et al. Длинная некодирующая РНК поддерживает активный хроматин для координации экспрессии гомеозисных генов. Природа. 2011; 472:120–124. [Бесплатная статья PMC] [PubMed] [Google Scholar]
15. Noller HF. Структура РНК: чтение рибосомы. Наука. 2005;309: 1508–1514. [PubMed] [Google Scholar]
Noller HF. Структура РНК: чтение рибосомы. Наука. 2005;309: 1508–1514. [PubMed] [Google Scholar]
16. Тур Н., Китинг К.С., Тейлор С.Д., Пайл А.М. Кристаллическая структура самосплайсированного интрона группы II. Наука. 2008; 320:77–82. [Бесплатная статья PMC] [PubMed] [Google Scholar]
17. Lanz RB, McKenna NJ, Onate SA, Albrecht U, Wong J, Tsai SY, Tsai MJ, O’Malley BW. Коактиватор стероидных рецепторов, SRA, функционирует как РНК и присутствует в комплексе SRC-1. Клетка. 1999; 97:17–27. [PubMed] [Google Scholar]
18. Hussein-Fikret S, Fuller PJ. Экспрессия корегуляторов ядерных рецепторов в стромальных и эпителиальных опухолях яичников. Мол. Клетка. Эндокринол. 2005;229: 149–160. [PubMed] [Google Scholar]
19. Lanz RB, Chua SS, Barron N, Söder BM, DeMayo F, O’Malley BW. Активатор РНК стероидных рецепторов стимулирует пролиферацию, а также апоптоз in vivo. Мол. Клетка. биол. 2003; 23:7163–7176. [Бесплатная статья PMC] [PubMed] [Google Scholar]
20. Leygue E, Dotzlaw H, Watson PH, Murphy LC. Экспрессия активатора РНК стероидного рецептора в опухолях молочной железы человека. Рак Рез. 1999;59:4190–4193. [PubMed] [Google Scholar]
Leygue E, Dotzlaw H, Watson PH, Murphy LC. Экспрессия активатора РНК стероидного рецептора в опухолях молочной железы человека. Рак Рез. 1999;59:4190–4193. [PubMed] [Google Scholar]
21. Murphy LC, Simon SL, Parkes A, Leygue E, Dotzlaw H, Snell L, Troup S, Adeyinka A, Watson PH. Измененная экспрессия корегуляторов рецептора эстрогена во время онкогенеза молочной железы человека. Рак Рез. 2000;60:6266–6271. [PubMed] [Академия Google]
22. Kawashima H, Takano H, Sugita S, Takahara Y, Sugimura K, Nakatani T. Новый белок-коактиватор стероидных рецепторов (SRAP) как альтернативная форма гена РНК-активатора стероидных рецепторов: экспрессия в клетках рака предстательной железы и усиление активности рецепторов андрогенов. Биохим. Дж. 2003; 369: 163–171. [Бесплатная статья PMC] [PubMed] [Google Scholar]
23. Emberley E, Huang GJ, Hamedani MK, Czosnek A, Ali D, Grolla A, Lu B, Watson PH, Murphy LC, Leygue E. Идентификация нового человека изоформы активаторов РНК стероидных рецепторов. Биохим. Биофиз. Рез. коммун. 2003;301:509–515. [PubMed] [Google Scholar]
Биохим. Биофиз. Рез. коммун. 2003;301:509–515. [PubMed] [Google Scholar]
24. Коулман К.М., Лам В., Джабер Б.М., Ланц Р.Б., Смит К.Л. Коактивация SRA альфа-рецептора эстрогена не зависит от фосфорилирования и усиливает активность агониста 4-гидрокситамоксифена. Биохим. Биофиз. Рез. коммун. 2004; 323:332–338. [PubMed] [Google Scholar]
25. Cavarretta IT, Mukopadhyay R, Lonard DM, Cowsert LM, Bennett CF, O’Malley BW, Smith CL. Снижение экспрессии коактиватора антисмысловыми олигодезоксинуклеотидами ингибирует транскрипционную активность ER-альфа и пролиферацию MCF-7. Мол. Эндокринол. 2002; 16: 253–270. [PubMed] [Академия Google]
26. Ши Ю., Даунс М., Се В., Као Х.И., Ордентлих П., Цай К.С., Хон М., Эванс Р.М. Sharp, индуцируемый кофактор, который объединяет репрессию и активацию ядерных рецепторов. Гены Дев. 2001; 15:1140–1151. [Бесплатная статья PMC] [PubMed] [Google Scholar]
27. Деблуа Г., Жигер В. Независимая от лиганда коактивация ERalpha AF-1 активатором РНК стероидного рецептора (SRA) посредством активации MAPK. Дж. Стероид Биохим. Мол. биол. 2003; 85: 123–131. [PubMed] [Google Scholar]
Дж. Стероид Биохим. Мол. биол. 2003; 85: 123–131. [PubMed] [Google Scholar]
28. Hatchell EC, Colley SM, Beveridge DJ, Epis MR, Stuart LM, Giles KM, Redfern AD, Miles LE, Barker A, MacDonald LM, et al. SLIRP, небольшой белок, связывающий SRA, является корепрессором ядерных рецепторов. Мол. Клетка. 2006; 22: 657–668. [PubMed] [Академия Google]
29. Курису Т., Танака Т., Исии Дж., Мацумура К., Сугимура К., Накатани Т., Кавасима Х. Экспрессия и функция активатора РНК стероидного рецептора человека в клетках рака предстательной железы: роль эндогенного белка hSRA в транскрипции, опосредованной рецептором андрогена. . Рак простаты Prostatic Dis. 2006; 9: 173–178. [PubMed] [Google Scholar]
30. Ланц Р.Б., Разани Б., Голдберг А.Д., О’Мэлли Б.В. Различные мотивы РНК важны для коактивации рецепторов стероидных гормонов активатором РНК стероидных рецепторов (SRA) Proc. Натл акад. науч. США. 2002;99:16081–16086. [Бесплатная статья PMC] [PubMed] [Google Scholar]
31. Zhao X, Patton JR, Davis SL, Florence B, Ames SJ, Spanjaard RA. Регуляция активности ядерных рецепторов псевдоуридинсинтазой посредством посттранскрипционной модификации РНК-активатора стероидных рецепторов. Мол. Клетка. 2004; 15: 549–558. [PubMed] [Google Scholar]
Zhao X, Patton JR, Davis SL, Florence B, Ames SJ, Spanjaard RA. Регуляция активности ядерных рецепторов псевдоуридинсинтазой посредством посттранскрипционной модификации РНК-активатора стероидных рецепторов. Мол. Клетка. 2004; 15: 549–558. [PubMed] [Google Scholar]
32. Xu B, Koenig RJ. РНК-связывающий домен в рецепторе гормона щитовидной железы усиливает активацию транскрипции. Дж. Биол. хим. 2004; 279:33051–33056. [PubMed] [Академия Google]
33. Сюй Б., Ян В.Х., Герин И., Ху К.Д., Хаммер Г.Д., Кениг Р.Дж. Dax-1 и активатор РНК рецептора стероидов (SRA) функционируют как коактиваторы транскрипции для стероидогенного фактора 1 в стероидогенезе. Мол. Клетка. биол. 2009; 29:1719–1734. [Бесплатная статья PMC] [PubMed] [Google Scholar]
34. Caretti G, Schiltz RL, Dilworth FJ, Di Padova M, Zhao P, Ogryzko V, Fuller-Pace FV, Hoffman EP, Tapscott SJ, Sartorelli V. РНК-хеликазы p68/p72 и некодирующая РНК SRA являются корегуляторами MyoD и дифференцировки скелетных мышц. Дев. Клетка. 2006; 11: 547–560. [PubMed] [Академия Google]
Дев. Клетка. 2006; 11: 547–560. [PubMed] [Академия Google]
35. Caretti G, Lei EP, Sartorelli V. Белки DEAD-box p68/p72 и некодирующий РНК-активатор стероидных рецепторов SRA: эклектичные регуляторы различных биологических функций. Клеточный цикл. 2007; 6: 1172–1176. [PubMed] [Google Scholar]
36. Yao H, Brick K, Evrard Y, Xiao T, Camerini-Otero RD, Felsenfeld G. Опосредование изоляции транскрипции CTCF с помощью DEAD-box РНК-связывающего белка p68 и активатора РНК стероидного рецептора СРА. Гены Дев. 2010;24:2543–2555. [Бесплатная статья PMC] [PubMed] [Google Scholar]
37. Ватанабе М., Янагисава Дж., Китагава Х., Такеяма К., Огава С., Арао Й., Сузава М., Кобаяши Й., Яно Т., Йошикава Х. и др. Подсемейство РНК-связывающих белков DEAD-box действует как коактиватор альфа-рецептора эстрогена через N-концевой домен активации (AF-1) с коактиватором РНК, SRA. EMBO J. 2001; 20: 1341–1352. [Бесплатная статья PMC] [PubMed] [Google Scholar]Retracted
38. Zhao X, Patton JR, Ghosh SK, Fischel-Ghodsian N, Shen L, Spanjaard RA. Pus3p- и Pus1p-зависимое псевдоуридилирование активатора РНК стероидных рецепторов контролирует функциональный переключатель, который регулирует передачу сигналов ядерных рецепторов. Мол. Эндокринол. 2007; 21: 686–69.9. [PubMed] [Google Scholar]
Pus3p- и Pus1p-зависимое псевдоуридилирование активатора РНК стероидных рецепторов контролирует функциональный переключатель, который регулирует передачу сигналов ядерных рецепторов. Мол. Эндокринол. 2007; 21: 686–69.9. [PubMed] [Google Scholar]
39. Chooniedass-Kothari S, Hamedani MK, Troup S, Hubé F, Leygue E. Белок-активатор РНК стероидного рецептора экспрессируется в тканях опухоли молочной железы. Междунар. Дж. Рак. 2006; 118:1054–1059. [PubMed] [Google Scholar]
40. Hubé F, Velasco G, Rollin J, Furling D, Francastel C. Белок-активатор РНК стероидного рецептора связывается с SRA-РНК-опосредованной активацией MyoD и дифференцировкой мышц и противодействует ей. Нуклеиновые Кислоты Res. 2011; 39: 513–525. [Бесплатная статья PMC] [PubMed] [Google Scholar]
41. Leygue E. Активатор РНК стероидных рецепторов (SRA1): необычные двусторонние генные продукты с подозрением на связь с раком молочной железы. Нуклеиновый Рецепт. Сигнал. 2007;5:e006. [Бесплатная статья PMC] [PubMed] [Google Scholar]
42. Cooper C, Vincett D, Yan Y, Hamedani MK, Myal Y, Leygue E. Активатор РНК стероидных рецепторов двугранная генетическая система: орел или решка? Биохимия. 2011; 93:1973–1980. [PubMed] [Google Scholar]
Cooper C, Vincett D, Yan Y, Hamedani MK, Myal Y, Leygue E. Активатор РНК стероидных рецепторов двугранная генетическая система: орел или решка? Биохимия. 2011; 93:1973–1980. [PubMed] [Google Scholar]
43. Hube F, Guo J, Chooniedass-Kothari S, Cooper C, Hamedani MK, Dibrov AA, Blanchard AA, Wang X, Deng G, Myal Y, et al. Альтернативный сплайсинг первого интрона РНК-активатора стероидных рецепторов (SRA) участвует в генерации кодирующих и некодирующих изоформ РНК в клеточных линиях рака молочной железы. ДНК-клеточная биол. 2006; 25: 418–428. [PubMed] [Академия Google]
44. Woese CR, Magrum LJ, Gupta R, Siegel RB, Stahl DA, Kop J, Crawford N, Brosius J, Gutell R, Hogan JJ, et al. Модель вторичной структуры бактериальной 16S рибосомной РНК: филогенетические, ферментативные и химические данные. Нуклеиновые Кислоты Res. 1980; 8: 2275–2293. [Бесплатная статья PMC] [PubMed] [Google Scholar]
45. Noller HF, Kop J, Wheaton V, Brosius J, Gutell RR, Kopylov AM, Dohme F, Herr W, Stahl DA, Gupta R, et al. Модель вторичной структуры 23S рибосомной РНК. Нуклеиновые Кислоты Res. 1981;9:6167–6189. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Модель вторичной структуры 23S рибосомной РНК. Нуклеиновые Кислоты Res. 1981;9:6167–6189. [Бесплатная статья PMC] [PubMed] [Google Scholar]
46. Коста М., Кристиан Э.Л., Мишель Ф. Дифференциальное химическое исследование саморасщепляющегося интрона группы II идентифицирует основания, участвующие в третичных взаимодействиях, и поддерживает альтернативную модель вторичной структуры домен В. РНК. 1998; 4: 1055–1068. [Бесплатная статья PMC] [PubMed] [Google Scholar]
47. Burgstaller P, Kochoyan M, Famulok M. Структурное зондирование и отбор повреждений цитруллин- и аргинин-специфических РНК-аптамеров определяют положения оснований, необходимые для связывания. Нуклеиновые Кислоты Res. 1995;23:4769–4776. [Бесплатная статья PMC] [PubMed] [Google Scholar]
48. Бениаминов А., Вестхоф Э., Крол А. Отличительные структуры между шимпанзе и человеком в некодирующей РНК мозга. РНК. 2008; 14:1270–1275. [PMC free article] [PubMed] [Google Scholar]
49. Новикова И.В., Хасан Б. Х., Мирзоян М.Г., Леонтис Н.Б. Разработка кооперативных комплексов текто-РНК с программируемой стехиометрией. Нуклеиновые Кислоты Res. 2011;39:2903–2917. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Х., Мирзоян М.Г., Леонтис Н.Б. Разработка кооперативных комплексов текто-РНК с программируемой стехиометрией. Нуклеиновые Кислоты Res. 2011;39:2903–2917. [Бесплатная статья PMC] [PubMed] [Google Scholar]
50. Watts JM, Dang KK, Gorelick RJ, Leonard CW, Bess JW, Jr, Swanstrom R, Burch CL, Weeks KM. Архитектура и вторичная структура всего генома РНК ВИЧ-1. Природа. 2009 г.;460:711–716. [Бесплатная статья PMC] [PubMed] [Google Scholar]
51. Regulski EE, Breaker RR. Поточный зондовый анализ рибопереключателей. Методы Мол. биол. 2008; 419: 53–67. [PubMed] [Google Scholar]
52. Уилкинсон К.А., Мерино Э.Дж., Уикс К.М. Селективное 2′-гидроксилацилирование, проанализированное путем удлинения праймера (SHAPE): количественный анализ структуры РНК с разрешением в один нуклеотид. Нац. протокол 2006; 1:1610–1616. [PubMed] [Google Scholar]
53. Mortimer SA, Weeks KM. Быстродействующий реагент для точного анализа вторичной и третичной структуры РНК с помощью SHAPE-химии. Варенье. хим. соц. 2007;129: 4144–4145. [PubMed] [Google Scholar]
Варенье. хим. соц. 2007;129: 4144–4145. [PubMed] [Google Scholar]
54. Vasa SM, Guex N, Wilkinson KA, Weeks KM, Giddings MC. ShapeFinder: программная система для высокопроизводительного количественного анализа информации о реактивности нуклеиновых кислот, разрешенной методом капиллярного электрофореза. РНК. 2008; 14:1979–1990. [Бесплатная статья PMC] [PubMed] [Google Scholar]
55. Thomas DJ, Rosenbloom KR, Clawson H, Hinrichs AS, Trumbower H, Raney BJ, Karolchik D, Barber GP, Harte RA, Hillman-Jackson J и др. . Проект ENCODE в Калифорнийском университете в Санта-Крус. Нуклеиновые Кислоты Res. 2007; 35:D663–D667. [Бесплатная статья PMC] [PubMed] [Google Scholar]
56. Хофакер И.Л., Фекете М., Штадлер П.Ф. Предсказание вторичной структуры выровненных последовательностей РНК. Дж. Мол. биол. 2002; 319:1059–1066. [PubMed] [Google Scholar]
57. Waterhouse AM, Procter JB, Martin DM, Clamp M, Barton GJ. Jalview Version 2 — редактор множественного выравнивания последовательностей и инструментальные средства анализа. Биоинформатика. 2009; 25:1189–1191. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Биоинформатика. 2009; 25:1189–1191. [Бесплатная статья PMC] [PubMed] [Google Scholar]
58. Gherghe CM, Shajani Z, Wilkinson KA, Varani G, Weeks KM. Сильная корреляция между химией SHAPE и обобщенным параметром порядка ЯМР (S2) в РНК. Варенье. хим. соц. 2008; 130:12244–12245. [Бесплатная статья PMC] [PubMed] [Google Scholar]
59. Мерино Э.Дж., Уилкинсон К.А., Кофлан Дж.Л., Уикс К.М. Анализ структуры РНК с разрешением в один нуклеотид с помощью селективного 2′-гидроксилацилирования и удлинения праймера (SHAPE) J. Am. хим. соц. 2005; 127:4223–4231. [PubMed] [Google Scholar]
60. Deigan KE, Li TW, Mathews DH, Weeks KM. Точное определение структуры РНК с помощью SHAPE. проц. Натл акад. науч. США. 2009; 106: 97–102. [Бесплатная статья PMC] [PubMed] [Google Scholar]
61. Тиджерина П., Мор С., Рассел Р. Отпечатки структурированных РНК и РНК-белковых комплексов с помощью DMS. Нац. протокол 2007; 2: 2608–2623. [Бесплатная статья PMC] [PubMed] [Google Scholar]
62. Локкард Р.Е., Кумар А. Картирование структуры тРНК в растворе с использованием двухцепочечной специфичной рибонуклеазы V1 из яда кобры. Нуклеиновые Кислоты Res. 1981; 9: 5125–5140. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Локкард Р.Е., Кумар А. Картирование структуры тРНК в растворе с использованием двухцепочечной специфичной рибонуклеазы V1 из яда кобры. Нуклеиновые Кислоты Res. 1981; 9: 5125–5140. [Бесплатная статья PMC] [PubMed] [Google Scholar]
63. Ziehler WA, Engelke DR. Исследование структуры РНК с помощью химических реагентов и ферментов. Курс. протокол нуклеиновая кислота хим. 2000; Глава 6: Раздел 6.1. [Бесплатная статья PMC] [PubMed] [Google Scholar]
64. Николс Н.М., Юэ Д. Рибонуклеазы. Курс. протокол Мол. биол. 2008; Глава 3: Раздел 3.13. [PubMed] [Академия Google]
65. Бен-Шем А., Гарро де Лубресс Н., Мельников С., Дженнер Л., Юсупова Г., Юсупов М. Структура эукариотической рибосомы при разрешении 3,0 А. Наука. 2011 Эп. [PubMed] [Google Scholar]
66. Dosztányi Z, Csizmok V, Tompa P, Simon I. IUPred: веб-сервер для предсказания изначально неструктурированных областей белков на основе расчетного содержания энергии. Биоинформатика. 2005;21:3433–3434. [PubMed] [Google Scholar]
67. Dosztányi Z, Csizmók V, Tompa P, Simon I. Парное содержание энергии, оцененное по аминокислотному составу, различает свернутые и неструктурированные белки. Дж. Мол. биол. 2005; 347: 827–839.. [PubMed] [Google Scholar]
Dosztányi Z, Csizmók V, Tompa P, Simon I. Парное содержание энергии, оцененное по аминокислотному составу, различает свернутые и неструктурированные белки. Дж. Мол. биол. 2005; 347: 827–839.. [PubMed] [Google Scholar]
68. Regad L, Martin J, Nuel G, Camproux AC. Добыча белковых петель с использованием структурного алфавита и статистической исключительности. Биоинформатика BMC. 2010;11:75. [Бесплатная статья PMC] [PubMed] [Google Scholar]
69. Cruz JA, Westhof E. Идентификация трехмерных структурных модулей в РНК на основе последовательностей с помощью RMDetect. Нац. Методы. 2011; 8: 513–521. [PubMed] [Google Scholar]
70. Chooniedass-Kothari S, Emberley E, Hamedani MK, Troup S, Wang X, Czosnek A, Hube F, Mutawe M, Watson PH, Leygue E. Активатор РНК стероидных рецепторов представляет собой первая функциональная РНК, кодирующая белок. ФЭБС лат. 2004; 566:43–47. [PubMed] [Академия Google]
71. Пейс Н.Р., Томас Б.К., Везе Ч.Р. Мир РНК. 2-е изд. Колд-Спринг-Харбор, Нью-Йорк: Лабораторное издательство Колд-Спринг-Харбор; 1999.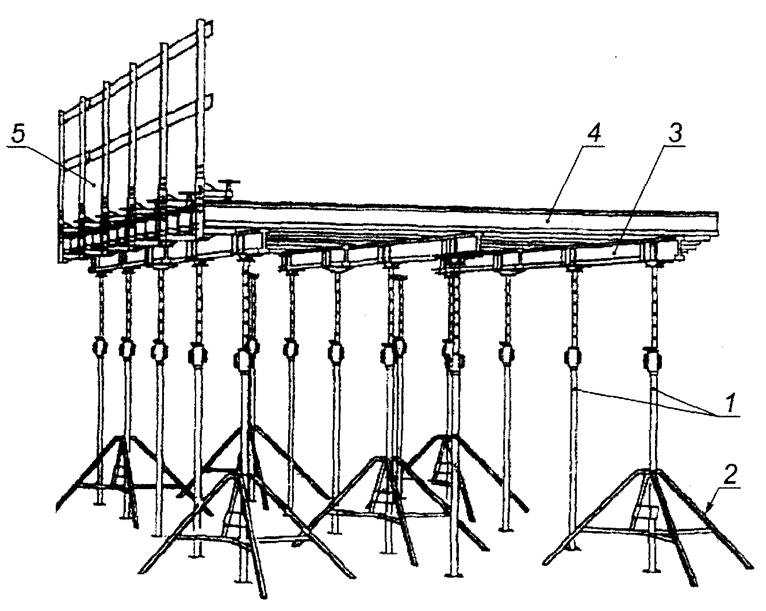 Исследование структуры, функции и истории РНК с помощью сравнительного анализа; п. 113. [Google Scholar]
Исследование структуры, функции и истории РНК с помощью сравнительного анализа; п. 113. [Google Scholar]
72. Мишель Ф., Вестхоф Э. Моделирование трехмерной архитектуры каталитических интронов группы I на основе сравнительного анализа последовательностей. Дж. Мол. биол. 1990; 216: 585–610. [PubMed] [Google Scholar]
73. Chooniedass-Kothari S, Vincett D, Yan Y, Cooper C, Hamedani MK, Myal Y, Leygue E. Белок, кодируемый активатором РНК функционального стероидного рецептора, является новым модулятором ER. альфа-транскрипционная активность. ФЭБС лат. 2010; 584:1174–1180. [PubMed] [Академия Google]
74. Струль К. Транскрипционный шум и достоверность инициации РНК-полимеразой II. Нац. Структура Мол. биол. 2007; 14:103–105. [PubMed] [Google Scholar]
75. Колли С.М., Лидман П.Дж. Активатор РНК стероидного рецептора — корегулятор ядерного рецептора с множеством партнеров: идеи и проблемы. Биохимия. 2011; 93:1966–1972. [PubMed] [Google Scholar]
76. Понтинг С.